如何使用Python做統計學上的回歸分析?
各位專家好: 若有一組數據型的資料, 想要對此資料進行回歸分析的話, 如畫出殘差圖、殘差Q-Q圖、Cook距離圖, 甚至是計算每個特徵的p-value來判定此特徵是否顯著的話, python是否有相關套件可以處理?或是否有相關程式碼或案例可供教學 感謝了~
回答列表
-
2022/03/12 上午 01:34王健安贊同數:0不贊同數:0留言數:1
Yaoga 您好, 殘差分析(Residual Analysis)有包含以下檢測項目,等會兒會一一講解虛無與對立假設、公式、判斷以及程式碼(以下舉例以線性迴歸為例)。 ```{python} # 引入套件 import numpy as np import pandas as pd import statsmodels.api as sm import statsmodels.stats.api as sms from statsmodels import stats import scipy # 匯入資料 row_data = pd.read_csv("boston-housing-dataset.csv") row_data.head() # 建立線性迴歸模型 linear_model = sm.OLS(endog = row_data["MEDV"], exog = row_data["LSTAT"]).fit() # 產生殘差值 res = row_data["MEDV"] - linear_model.fittedvalues ``` 1. 殘差呈現常態分佈:可使用 Q-Q plot 繪圖或是 Shapiro test 檢定殘差是否呈現常態分佈。 $$ \\ \begin{cases} H_{0}: The\ residual\ is\ normal\ distribution. \\ H_{1}: The\ residual\ is\ not\ normal\ distribution. \\ \end{cases} \\ $$ ```{python} # Normalty scipy.stats.shapiro(res) ``` 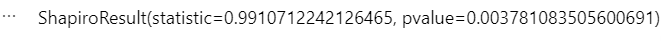 從結果可知 p-value = 0.003 < 0.05,不拒絕虛無假設,代表殘差值不呈現常態分佈,需要針對依變項進行資料轉換。 2. 殘差變異數一致性:使用 Breusch-Pagan Test 檢測殘差變異數是否一致 $$ \begin{cases} H_{0}: \sigma^{1} = \sigma^{2} = ... = \sigma^{n} \\ H_{1}: \sigma^{i} \not= \sigma^{j} \\ \end{cases} $$ ```{python} # Variance Hemogeneity sms.het_breuschpagan(res, linear_model.model.exog) ``` 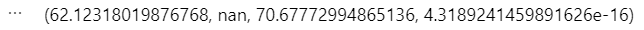 結果顯示 p-value = 4.31E-16 < 0.05,拒絕虛無假設,代表殘差變異數不一致。 3. 每個殘差之間互為獨立:可使用 Durban-Watson test 檢定每個殘差之間是否獨立。 $$ \begin{cases} H_{0}: The\ errors\ is\ independent. \\ H_{1}: The\ errors\ is\ not\ independent. \\ \end{cases} $$ ```{python} # Sample Inependence tstatistic = stats.stattools.durbin_watson(res) # 計算出 T 統計量 tpvalue = scipy.stats.t.sf(tstatistic, 504) # 將統計量轉換成 p-value print(tstatistic, tpvalue) ``` 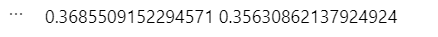 結果顯示 p-value = 0.356 > 0.05,接受虛無假設,代表每個殘差之間互為獨立。 4. 找出影響點:可使用 Cook's Distance ```{python} # Use Cook's Distance to find influence plot fig = sm.graphics.influence_plot(linear_model, criterion = "cooks") fig.tight_layout(pad = 1.0) ``` 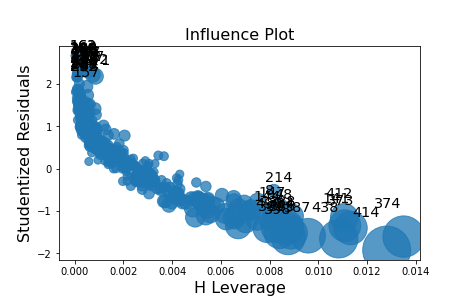 參考資料 https://towardsdatascience.com/how-to-use-residual-plots-for-regression-model-validation-c3c70e8ab378 https://www.statsmodels.org/devel/generated/statsmodels.stats.stattools.durbin_watson.html https://www.statology.org/breusch-pagan-test-python/
